Comment j’ai fait séquencer mon ADN, partie 2
jeudi 5 février 2015 5 commentaires
Suite de la première partie.
A partir du moment où on reçoit ses résultats de séquençage, s’ouvrent de très nombreuses possibilités d’analyse et d’interprétation. La limite à ce que l’on peut faire tient surtout à quel niveau de vie privée on souhaite maintenir (ou pas). Le site 23andyou donne un bon aperçu des options disponibles pour les clients de 23andme, et la plupart de ces options marchent aussi pour les clients des autres séquenceurs grand public.
Bienvenue à Gattaca:
Comme mentionné dans la partie 1, 23andme ne fournit plus d’informations générales de santé mais seulement des détails d’origine ancestrale (proportion d’ADN issu de néanderthal, haplotypes mitochondrial et Y, composition ethnique à la grosse louche) et généalogique (pays d’origine, cousins génétiques plus ou moins distants connus dans leur base). Avec des outils de tierce partie il est possible de compléter et améliorer ces résultats d’origine, ainsi qu’obtenir des informations de santé. On peut ainsi faire appel à GeneKnot (gratuit et potentiellement anonyme) ou à Promethease (l’analyse coûte 5 dollars US, payables par carte bleue ou Paypal, ce qui rend l’anonymat à peu près impossible – notez qu’ils acceptaient les Bitcoins, et prévoient de réinstaurer cette capacité).
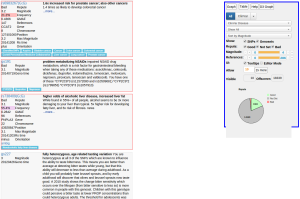
Comment se présente mon rapport de Promethease (exclusivement en anglais). Effectivement j’apprécie mieux la bière et les choux de Bruxelles que quand j’étais enfant…
Les deux outils donnent une liste de vos gènes qui modifient potentiellement vos risques pour tout un tas de conditions médicales (DMLA, Alzheimer, diabète, asthme, certains cancers, ou encore.. les taches de rousseur) mais ils sont assez « arides ». GeneKnot donne un rapport condensé, tandis que Promethéase sort tout ce qu’il est possible de produire sur toutes les mutations que vous avez (même celles en un seul exemplaire) et pour lesquelles il existe une publication répertoriée dans sa base de données (SNPedia). Il vaut mieux avoir de bonnes connaissances en recherche pour interpréter les résultats car, généralement, qu’on ait constaté une augmentation de 20 ou 30% de risque d’avoir une maladie statistiquement associée à un gène particulier est très insuffisant pour croire à un lien de cause à effet possible entre le gène et la maladie. Et savoir que l’on a 10% de risque en moins que la moyenne de développer un diabète de type 2 n’est en rien une façon de s’excuser de reprendre une part de gâteau au dessert… Pour le dire simplement, la plus grande part des rapports médicaux que sortent ce genre d’outil est inutile voire trompeur, à moins d’avoir confirmation d’être porteur ou malade de quelque chose de sérieux comme une hémochromatose ou une dystrophie. C’est d’ailleurs ce type de constat qui avait à l’origine motivé la FDA pour prohiber ce service chez 23andme.
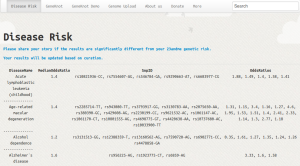
Le rapport GeneKnot est encore plus sec. Non je n’ai pas eu de leucémie, merci. D’ailleurs passer d’1 chance sur 6600 à 1 chance sur 4500 de l’avoir n’est pas vraiment terrifiant.
Bref, les informations dévoilées apportent un éclairage intéressant sur son propre caractère et performances sportives, ou éventuellement peuvent donner des mises en garde sur sa santé à venir, mais il ne vaut mieux pas les considérer comme un prérequis pour prendre soin de soi, ni s’imaginer qu’obtenir ces informations sera un déclencheur suffisant pour vous motiver à changer d’hygiène de vie. Pour illustrer, j’ai déjà été obèse et je savais déjà d’expérience que l’alcool me rend très vite malade, donc faute de me découvrir malade de quelque chose d’inattendu et bien connu médicalement, ces résultats n’ont rien changé pour moi.
Ethnicité et approximations:
Vient ensuite la partie la plus amusante car tellement entachée de drama: l’analyse des origines ancestrales et ethniques. Comme mentionné dans la partie 1, les analyses proposées par 23andme ont des biais qui rendent les résultats approximatifs voire faux. Dans mon cas, à quel point ? C’est ce que j’ai voulu savoir…
Parmi les outils respectant le secret des données, il y a par exemple Interpretome, qui s’exécute dans votre navigateur et donc ne requiert pas de transmettre votre génome entier à un tiers. Sinon il y a GEDmatch qui rend aisée l’analyse ethnique et ancestrale en plus de proposer une recherche de parenté avec les autres profils, utilisable de manière suffisamment anonyme (il attribue un identifiant numérique au génome). On peut tester le fonctionnement de ces outils gratuits sur des génomes publics pour mieux comprendre leur utilité: passer son génome à la moulinette informatique de tests conçus pour l’anthropologie génétique, a priori plus précis et tenus mieux à jour que ceux des entreprises commerciales.
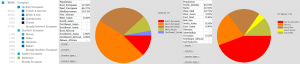
A gauche, ce que me dit 23andme, et à droite mes résultats Dodecad v3 et Eurogenes K13.
Oups, il y a comme un bug, mes ancêtres polonais et pachtoune ont été escamotés…
Parmi les analyses les plus intéressantes que l’on peut faire avec son génome, il y a les analyses en composantes principales: celles-ci consistent à pondérer de manière très compliquée plusieurs (dizaines voire centaines de) milliers de SNPs afin de positionner les individus sur des axes, en choisissant soigneusement les pondérations appliquées pour maximiser les distinctions qu’il est possible de faire. Plus simplement, cela permet de mesurer votre distance à certaines populations de référence, en se basant sur la génétique et les mathématiques (plutôt que, comme peuvent le faire les plus bornés des racistes, sur des critères grossiers et trompeurs comme la couleur de la peau, la taille du nez ou l’accent). Avec 2 pondérations (=composantes principales), on obtient 2 axes permettant de tracer une carte en 2 dimensions des populations étudiées. Une CP en plus, et cela devient une carte en 3D. Ordinairement on génère une dizaine de ces axes pour bien mettre en évidence les différences et similarités entre les populations, ainsi que distinguer de possibles sous-populations internes.

Et hop, comment se découvrir subitement d’origine proche-orientale plutôt qu’européenne.
Il y a plusieurs projets de référencement des populations, à l’échelle mondiale (Human Genome Diversity Project, HapMap) ou régionale (Harappa pour l’Asie, EthioHelix pour l’Afrique, RefPop pour l’Europe, Magnus Ducatus Lituaniae Project pour l’Eurasie…), permettant de procéder en plusieurs étapes et d’affiner des origines mixtes, et ainsi obtenir des compositions ancestrales beaucoup plus précises.
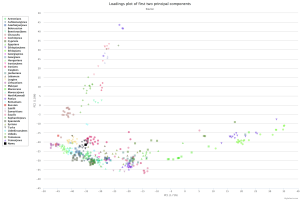
Ma part moyen-orientale serait turco-perse. Peut-être kurde ?
De même sur la détermination des haplotypes (« cartes d’identité » des ADN mitochondrial – hérité exclusivement de votre mère – et chromosome Y – hérité exclusivement de votre père), il est utile de refaire l’analyse séparément (par exemple à l’aide des outils d’ISOGG ou OpenSNP). Dans mon cas, 23andme donnait un résultat très proche mais pas tout à fait exact. Chaque série de mutations spécifiques présentes dans ces deux sources correspond à une histoire de migration ancestrale particulière, intimement liée à l’évolution de notre espèce. Ces données ont une utilité généalogique comme anthropologique car elles définissent de manière très précise et sur des milliers d’années vos filiations patri et matrilinéaires, et les mouvements de populations du passé lointain.
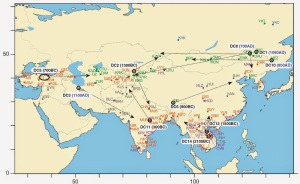
Une reconstitution des mouvements de population basée sur les mutations du chromosome Y chez les descendants
European Journal of Human Genetics: Y-chromosome descent clusters and male differential reproductive success: young lineage expansions dominate Asian pastoral nomadic populations ; Graphique 4
Patricia Balaresque et al.
Attention quand même, si vous multipliez les tests sur vos parents proches: se découvrir d’une origine patrilinéaire différente de son géniteur supposé peut facilement mettre en péril la cohésion d’une famille… En la matière, l’analyse génétique a énormément à offrir, mais avant de commencer à creuser il vaut mieux être sûr de vouloir en savoir autant. Dans la partie finale, je parlerai de généalogie proche, de transmission de l’ADN, et de fragmentation…
re privacy, promethease took bitcoin payments for a while, and will again. sorry it’s not currently available.
Thanks for that detail, I’ve updated the story.
Ping: Comment j’ai fait séquencer mon ADN (2) | Contrepoints
Pour référence, à l’heure actuelle 23andme utilise toujours des versions d’arbre d’haplogroupes Y et mitochondrial datant de fin 2009 (ISOGG 2009 pour le Y, Phylotree Build 7 de 2009 pour mt).
Bonjour,
Voilà des articles très intéressants !
Je n’ai pas trouvé la partie finale. Pourriez-vous indiquer où la trouver ?
Merci d’avance.